













| General Information: |
|
| Name(s) found: |
ISW1_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 1129 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MAYMLAIANF HFFKFYTRMR KKHENNSCNE KDKDENLFKI ILAIFLQEKK KYDCISSGSI 60
61 MTASEEYLEN LKPFQVGLPP HDPESNKKRY LLKDANGKKF DLEGTTKRFE HLLSLSGLFK 120
121 HFIESKAAKD PKFRQVLDVL EENKANGKGK GKHQDVRRRK TEHEEDAELL KEEDSDDDES 180
181 IEFQFRESPA YVNGQLRPYQ IQGVNWLVSL HKNKIAGILA DEMGLGKTLQ TISFLGYLRY 240
241 IEKIPGPFLV IAPKSTLNNW LREINRWTPD VNAFILQGDK EERAELIQKK LLGCDFDVVI 300
301 ASYEIIIREK SPLKKINWEY IIIDEAHRIK NEESMLSQVL REFTSRNRLL ITGTPLQNNL 360
361 HELWALLNFL LPDIFSDAQD FDDWFSSEST EEDQDKIVKQ LHTVLQPFLL RRIKSDVETS 420
421 LLPKKELNLY VGMSSMQKKW YKKILEKDLD AVNGSNGSKE SKTRLLNIMM QLRKCCNHPY 480
481 LFDGAEPGPP YTTDEHLVYN AAKLQVLDKL LKKLKEEGSR VLIFSQMSRL LDILEDYCYF 540
541 RNYEYCRIDG STAHEDRIQA IDDYNAPDSK KFVFLLTTRA GGLGINLTSA DVVVLYDSDW 600
601 NPQADLQAMD RAHRIGQKKQ VKVFRLVTDN SVEEKILERA TQKLRLDQLV IQQNRTSLKK 660
661 KENKADSKDA LLSMIQHGAA DVFKSGTSTG SAGTPEPGSG EKGDDIDLDE LLLKSENKTK 720
721 SLNAKYETLG LDDLQKFNQD SAYEWNGQDF KKKIQRDIIS PLLLNPTKRE RKENYSIDNY 780
781 YKDVLNTGRS STPSHPRMPK PHVFHSHQLQ PPQLKVLYEK ERMWTAKKTG YVPTMDDVKA 840
841 AYGDISDEEE KKQKLELLKL SVNNSQPLTE EEEKMKADWE SEGFTNWNKL EFRKFITVSG 900
901 KYGRNSIQAI ARELAPGKTL EEVRAYAKAF WSNIERIEDY EKYLKIIENE EEKIKRVKMQ 960
961 QEALRRKLSE YKNPFFDLKL KHPPSSNNKR TYSEEEDRFI LLMLFKYGLD RDDVYELVRD1020
1021 EIRDCPLFEL DFYFRSRTPV ELARRGNTLL QCLEKEFNAG IVLDDATKDR MKKEDENGKR1080
1081 IREEFADQTA NEKENVDGVE SKKAKIEDTS NVGTEQLVAE KIPENETTH |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
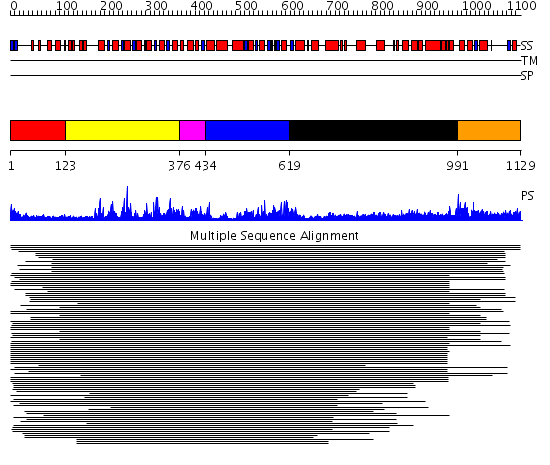
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..122] | 1.231996 | View MSA. Confident ab initio structure predictions are available. | |
| 2 | View Details | [123..375] | 4.69897 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 3 | View Details | [376..433] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [434..618] | 11.154902 | Putative DEAD box RNA helicase | |
| 5 | View Details | [619..990] | 3.688996 | View MSA. No confident structure predictions are available. | |
| 6 | View Details | [991..1129] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)