













| General Information: |
|
| Name(s) found: |
CHL1_YEAST
[Swiss-Prot]
|
| Description(s) found:
Found 2 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 861 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MDKKEYSETF YHPYKPYDIQ VQLMETVYRV LSEGKKIAIL ESPTGTGKTL SLICATMTWL 60
61 RMNKADIFTR METNIKTNED DSENLSDDEP DWVIDTYRKS VLQEKVDLLN DYEKHLNEIN 120
121 TTSCKQLKTM CDLDKEHGRY KSVDPLRKKR KGARHLDVSL EEQDFIPRPY ESDSENNDTS 180
181 KSTRGGRISD KDYKLSELNS QIITLLDKID GKVSRDPNNG DRFDVTNQNP VKIYYASRTY 240
241 SQLGQFTSQL RLPSFPSSFR DKVPDEKVKY LPLASKKQLC INPKVMKWKT LEAINDACAD 300
301 LRHSKEGCIF YQNTNEWRHC PDTLALRDMI FSEIQDIEDL VPLGKSLGIC PYYASREALP 360
361 IAEVVTLPYQ YLLSESTRSS LQINLENSIV IIDEAHNLIE TINSIYSSQI SLEDLKNCHK 420
421 GIVTYFNKFK SRLNPGNRVN LLKLNSLLMT LIQFIVKNFK KIGQEIDPND MFTGSNIDTL 480
481 NIHKLLRYIK VSKIAYKIDT YNQALKEEES SKNENPIKET HKKSVSSQPL LFKVSQFLYC 540
541 LTNLTSEGQF FFEKNYSIKY MLLEPSKPFE SILNQAKCVV LAGGTMEPMS EFLSNLLPEV 600
601 PSEDITTLSC NHVIPKENLQ TYITNQPELE FTFEKRMSPS LVNNHLFQFF VDLSKAVPKK 660
661 GGIVAFFPSY QYLAHVIQCW KQNDRFATLN NVRKIFYEAK DGDDILSGYS DSVAEGRGSL 720
721 LLAIVGGKLS EGINFQDDLC RAVVMVGLPF PNIFSGELIV KRKHLAAKIM KSGGTEEEAS 780
781 RATKEFMENI CMKAVNQSVG RAIRHANDYA NIYLLDVRYN RPNFRKKLSR WVQDSINSEH 840
841 TTHQVISSTR KFFSMRSLNS R |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
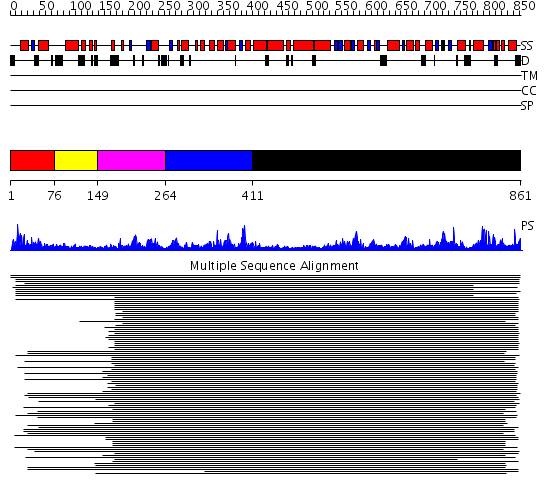
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..75] | 2.07099 | View MSA. Confident ab initio structure predictions are available. | |
| 2 | View Details | [76..148] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [149..263] | 3.522879 | Putative DEAD box RNA helicase | |
| 4 | View Details | [264..410] | N/A | No confident structure predictions are available. | |
| 5 | View Details | [411..861] | 13.187997 | View MSA. No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.96 |
Source: Reynolds et al. (2008)