













| General Information: |
|
| Name(s) found: |
RAD3_YEAST
[Swiss-Prot]
|
| Description(s) found:
Found 2 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 778 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MKFYIDDLPV LFPYPKIYPE QYNYMCDIKK TLDVGGNSIL EMPSGTGKTV SLLSLTIAYQ 60
61 MHYPEHRKII YCSRTMSEIE KALVELENLM DYRTKELGYQ EDFRGLGLTS RKNLCLHPEV 120
121 SKERKGTVVD EKCRRMTNGQ AKRKLEEDPE ANVELCEYHE NLYNIEVEDY LPKGVFSFEK 180
181 LLKYCEEKTL CPYFIVRRMI SLCNIIIYSY HYLLDPKIAE RVSNEVSKDS IVIFDEAHNI 240
241 DNVCIESLSL DLTTDALRRA TRGANALDER ISEVRKVDSQ KLQDEYEKLV QGLHSADILT 300
301 DQEEPFVETP VLPQDLLTEA IPGNIRRAEH FVSFLKRLIE YLKTRMKVLH VISETPKSFL 360
361 QHLKQLTFIE RKPLRFCSER LSLLVRTLEV TEVEDFTALK DIATFATLIS TYEEGFLLII 420
421 EPYEIENAAV PNPIMRFTCL DASIAIKPVF ERFSSVIITS GTISPLDMYP RMLNFKTVLQ 480
481 KSYAMTLAKK SFLPMIITKG SDQVAISSRF EIRNDPSIVR NYGSMLVEFA KITPDGMVVF 540
541 FPSYLYMESI VSMWQTMGIL DEVWKHKLIL VETPDAQETS LALETYRKAC SNGRGAILLS 600
601 VARGKVSEGI DFDHQYGRTV LMIGIPFQYT ESRILKARLE FMRENYRIRE NDFLSFDAMR 660
661 HAAQCLGRVL RGKDDYGVMV LADRRFSRKR SQLPKWIAQG LSDADLNLST DMAISNTKQF 720
721 LRTMAQPTDP KDQEGVSVWS YEDLIKHQNS RKDQGGFIEN ENKEGEQDED EDEDIEMQ |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
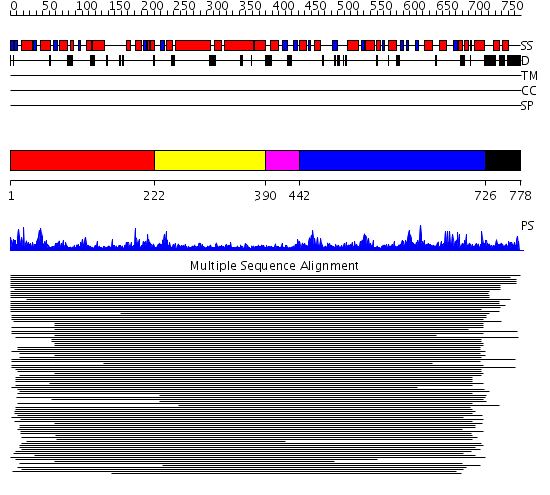
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..221] | 39.0 | Putative DEAD box RNA helicase | |
| 2 | View Details | [222..389] | 39.0 | Putative DEAD box RNA helicase | |
| 3 | View Details | [390..441] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [442..725] | 9.08 | Initiation factor 4a | |
| 5 | View Details | [726..778] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)