













| General Information: |
|
| Name(s) found: |
gi|168267318
[NCBI NR]
|
| Description(s) found: |
|
| Organism: | synthetic construct |
| Length: | 747 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSKLKSSESV RVVVRCRPMN GKEKAASYDK VVDVDVKLGQ VSVKNPKGTA HEMPKTFTFD 60
61 AVYDWNAKQF ELYDETFRPL VDSVLQGFNG TIFAYGQTGT GKTYTMEGIR GDPEKRGVIP 120
121 NSFDHIFTHI SRSQNQQYLV RASYLEIYQE EIRDLLSKDQ TKRLELKERP DTGVYVKDLS 180
181 SFVTKSVKEI EHVMNVGNQN RSVGATNMNE HSSRSHAIFV ITIECSEVGL DGENHIRVGK 240
241 LNLVDLAGSE RQAKTGAQGE RLKEATKINL SLSALGNVIS ALVDGKSTHI PYRDSKLTRL 300
301 LQDSLGGNAK TVMVANVGPA SYNVEETLTT LRYANRAKNI KNKPRVNEDP KDALLREFQE 360
361 EIARLKAQLE KRSIGRRKRR EKRREGGGSG GGGEEEEEEG EEGEEEGDDK DDYWREQQEK 420
421 LEIEKRAIVE DHSLVAEEKM RLLKEKEKKM EDLRREKDAA EMLGAKIKAM ESKLLVGGKN 480
481 IVDHTNEQQK ILEQKRQEIA EQKRREREIQ QQMESRDEET LELKETYSSL QQEVDIKTKK 540
541 LKKLFSKLQA VKAEIHDLQE EHIKERQELE QTQNELTREL KLKHLIIENF IPLEEKSKIM 600
601 NRAFFDEEED HWKLHPITRL ENQQMMKRPV SAVGYKRPLS QHARMSMMIR PEARYRAENI 660
661 VLLELDMPSR TTRDYEGPAI APKVQAALDA ALQDEDEIQV DASSFESTAN KKSKARPKSG 720
721 RKSGSSSSSS GTPASQLYPQ SRGLVPK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
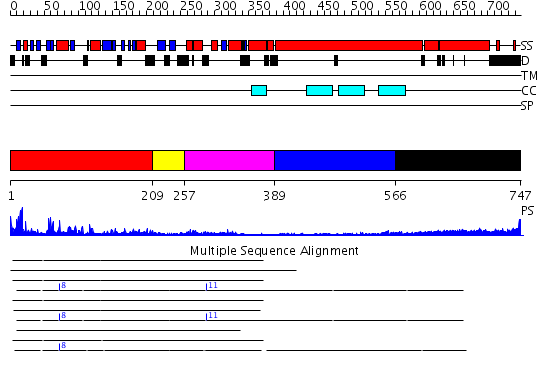
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..208] | 71.154902 | Kinesin | |
| 2 | View Details | [209..256] | 3.17 | Kinesin | |
| 3 | View Details | [257..388] | 8.522879 | KINESIN (DIMERIC) FROM RATTUS NORVEGICUS | |
| 4 | View Details | [389..565] | 2.24 | Ribonuclease domain of colicin E3; Colicin E3 translocation domain; Colicin E3 receptor domain | |
| 5 | View Details | [566..747] | 5.522879 | No description for 2i1jA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)