













| General Information: |
|
| Name(s) found: |
fft2 /
SPCC1235.05c
[Sanger Pombe]
|
| Description(s) found:
Found 19 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 1284 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IC nuclear chromatin [NAS] cytosol [IDA chromatin remodeling complex [NAS |
| Biological Process: |
chromatin remodeling
[NAS chromosome organization [ISS |
| Molecular Function: |
ATP binding
[ISS DNA binding [IEA] ATP-dependent DNA helicase activity [ISS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MLPYNSNYLS DNGKRKFSDE NPQSEVYGSS QTGLPSSYGN PQSYGTPPVQ QSSAMYGVNN 60
61 SMGGGMYNTS ENTQFMNTDY SQTSSYASTP MSNAYSRDAP AAINNNFGYS YVGQSSQPVP 120
121 SYNPLPSYNT ASLPNAGIPA AMPGMPSGYP GTVPIPQGGY NAHYSSPYNN GYPIGAVNPT 180
181 SAIPAQPPAQ PVNNVLPSYV RSNSRSSARS TARSAPRSTQ RSRSSSANPV TTPPVNNTLL 240
241 TPPAPPVELP PVTTTSPNAI IRSVQWIRSF VPQAPIHQVI NTLAQTKWDE TAALSILSQK 300
301 YLSCDLGIPI QEHKRFKQSP VASNMPTYGS SNRTVQSQKR SIRDKYIQMP NDSTQASLMP 360
361 SYTRKTSNAS KKLTTEEDEF YDSEEEPEAI VHRDTSALER TVLNFINSST AKELSDTASC 420
421 PLSHSKLLLE HRPFQTLAEA CIIKHPDDVP SKPGRRGRRR EKNPMGQKIV NACMETMEGY 480
481 YAIDNLIAKC EFLGNRISKG MASWGIKLEM SNGELNIVDM ESVPTEAADN SDFPKFVTEQ 540
541 PKTLASDVQL KSYQLVGVNW LHLLYQQKLS GILADEMGLG KTCQVVAFFA LLLEQGHHGP 600
601 HLVVVPSSTL ENWLRELARF CPSLRVEPYY GSQQERANIR EAIEENEIKY DILVTTYQLA 660
661 TNNKEDRSFL KHQNFDVCVY DEGHYLKNRM SERYKHLMNL NANFRLLLTG TPLQNNLKEL 720
721 VSLLAFILPN MFDSDMDDLD VIFKAKPTAD ADIEQALLSK QRISRAKTMM TPFVLRRRKN 780
781 QVLNDLPKKT QIIEHCKLSE NQLEIYNRYA ALQKNQQLRR DDKRNKRSKN DEESDGKSLS 840
841 AGHVLMQLRK AANHALLFRK FYDDEKLKQM AKDIMQEEQY KNANEQYIYE DMEVMSDFEL 900
901 HRLCRSFPTL QSYTLKDDPW MDSGKIRVLK ELLPKMKEEG SRILLFSQFT QMLDILEQVL 960
961 DTLKISYVRL DGSTQVEVRQ DIIDQFHKEE DVTVFLLSTK AGGFGINLAC ANVVILYDCS1020
1021 YNPFDDLQAE DRAHRVGQVR EVTVIRLITD NTIEEYIQKL ANTKLALDMS LSSDGKDREE1080
1081 IGERLVQDML DEENNGNNTK PEITGNESDG EFKVSSSNNS KQTDAEETNT GVPLEGSQPN1140
1141 SVEKTDLADG DEKANIKTEM KSETVEGDNK ELRETMKGEN VQTDSNAAVP SSKSSTEEPN1200
1201 ESVLSGHLDL DTEASPVVST IEKTTKGDVS VTEEQQSANI DGQLEKPEIE ESKKPDVLNQ1260
1261 VSLSIEEEKP KNKESEVDNN AAKD |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
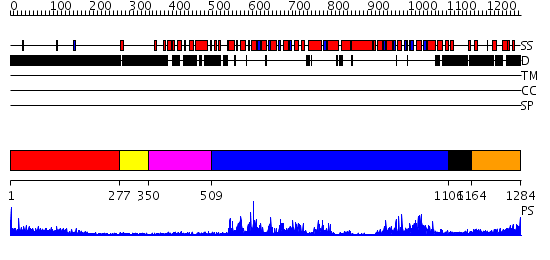
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..276] | 4.221849 | Sec24 | |
| 2 | View Details | [277..349] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [350..508] | 2.41 | Architecture of the photosynthetic oxygen evolving center | |
| 4 | View Details | [509..1105] | 125.0 | Structure of the SWI2/SNF2 chromatin remodeling domain of eukaryotic Rad54 | |
| 5 | View Details | [1106..1163] | 7.39794 | Crystal structure of Escherichia coli transcription-repair coupling factor | |
| 6 | View Details | [1164..1284] | 7.0 | nucleosome recognition module of ISWI ATPase |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)