













| General Information: |
|
| Name(s) found: |
FBpp0302531
[FlyBase]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 547 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSFMNPVDMV DEDAADLQFP KVEPLGPQVK RFIHAHQNEH TRNSMVLYNP HTLHRYLEEM 60
61 EEKTRDQNSS VPSATKDANR CATCRCSLTE PYIKCSECLD TLLCLQCFSR GKEAFSHRNN 120
121 HAYIIVRDNI QVFADEPHWT ARDERILLKT LRTHGYGNWE AVSQALDQRH EPAEVRRHYH 180
181 DCYFGGIFER LLNLKHARDS YVPERMPYVF KMRSLDPPRH DDIASMQFRL SAGYRCARGD 240
241 FDTPYDTSAE SLLSIMVDHR GRDDDNEASE SEFEREVTEE LQLGLVRAYN NRLRERQRRY 300
301 KIMRQHGLIM PNRTVSWISK YVHAFGSDAS CMRFLGFMQI CPDPIKFDML LESLRYYREL 360
361 HSQLHKLYDL REHGVRTLSG AKLYARLSKE RQQAQRDYSR LKQTDAFDWQ QLVQHYESNR 420
421 SGDPGPLAIN SKLYVMNTRR KASPIEIGGG KHFTHCLTPT EYNFSLIPDL PGYSKLDDGE 480
481 RKLCSVARLV PQSYLDYKNQ LVTEQAKLGY LRLADARRLI KIDVNKTRQI YDFLLEHGHI 540
541 SRPPSYG |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
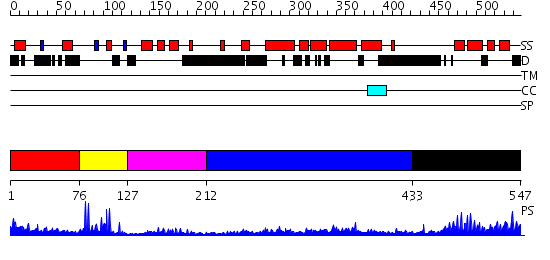
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..75] | 7.69897 | Dystrophin | |
| 2 | View Details | [76..126] | 2.28 | No description for 2fc7A was found. | |
| 3 | View Details | [127..211] | 1.296995 | View MSA. No confident structure predictions are available. | |
| 4 | View Details | [212..432] | 3.215992 | View MSA. No confident structure predictions are available. | |
| 5 | View Details | [433..547] | 27.39794 | Structural and functional analysis of ada2 alpha swirm domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)