













| General Information: |
|
| Name(s) found: |
MTR4_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 1073 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MDSTDLFDVF EETPVELPTD SNGEKNADTN VGDTPDHTQD KKHGLEEEKE EHEENNSENK 60
61 KIKSNKSKTE DKNKKVVVPV LADSFEQEAS REVDASKGLT NSETLQVEQD GKVRLSHQVR 120
121 HQVALPPNYD YTPIAEHKRV NEARTYPFTL DPFQDTAISC IDRGESVLVS AHTSAGKTVV 180
181 AEYAIAQSLK NKQRVIYTSP IKALSNQKYR ELLAEFGDVG LMTGDITINP DAGCLVMTTE 240
241 ILRSMLYRGS EVMREVAWVI FDEVHYMRDK ERGVVWEETI ILLPDKVRYV FLSATIPNAM 300
301 EFAEWICKIH SQPCHIVYTN FRPTPLQHYL FPAHGDGIYL VVDEKSTFRE ENFQKAMASI 360
361 SNQIGDDPNS TDSRGKKGQT YKGGSAKGDA KGDIYKIVKM IWKKKYNPVI VFSFSKRDCE 420
421 ELALKMSKLD FNSDDEKEAL TKIFNNAIAL LPETDRELPQ IKHILPLLRR GIGIHHSGLL 480
481 PILKEVIEIL FQEGFLKVLF ATETFSIGLN MPAKTVVFTS VRKWDGQQFR WVSGGEYIQM 540
541 SGRAGRRGLD DRGIVIMMID EKMEPQVAKG MVKGQADRLD SAFHLGYNMI LNLMRVEGIS 600
601 PEFMLEHSFF QFQNVISVPV MEKKLAELKK DFDGIEVEDE ENVKEYHEIE QAIKGYREDV 660
661 RQVVTHPANA LSFLQPGRLV EISVNGKDNY GWGAVVDFAK RINKRNPSAV YTDHESYIVN 720
721 VVVNTMYIDS PVNLLKPFNP TLPEGIRPAE EGEKSICAVI PITLDSIKSI GNLRLYMPKD 780
781 IRASGQKETV GKSLREVNRR FPDGIPVLDP VKNMKIEDED FLKLMKKIDV LNTKLSSNPL 840
841 TNSMRLEELY GKYSRKHDLH EDMKQLKRKI SESQAVIQLD DLRRRKRVLR RLGFCTPNDI 900
901 IELKGRVACE ISSGDELLLT ELIFNGNFNE LKPEQAAALL SCFAFQERCK EAPRLKPELA 960
961 EPLKAMREIA AKIAKIMKDS KIEVVEKDYV ESFRHELMEV VYEWCRGATF TQICKMTDVY1020
1021 EGSLIRMFKR LEELVKELVD VANTIGNSSL KEKMEAVLKL IHRDIVSAGS LYL |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
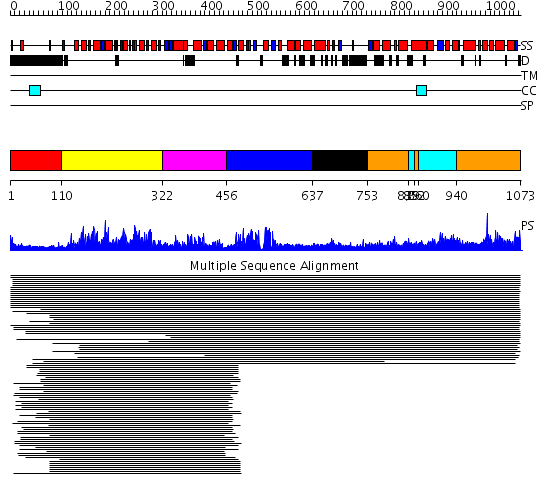
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..109] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [110..321] | 89.522879 | Putative DEAD box RNA helicase | |
| 3 | View Details | [322..455] | 89.522879 | Putative DEAD box RNA helicase | |
| 4 | View Details | [456..636] | 25.39794 | Initiation factor 4a | |
| 5 | View Details | [637..752] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [753..838] [852..859] [940..1073] |
8.05 | (+)-bornyl diphosphate synthase | |
| 7 | View Details | [839..851] [860..939] |
8.05 | (+)-bornyl diphosphate synthase |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)