













| General Information: |
|
| Name(s) found: |
KIP3_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 805 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MNVPETRQSS IVVAIRVRPF TSMEKTRLVN EASGAEANFP GLGDSSLILP MSNNSDSDID 60
61 IDAEEGSTRS KRNSLLRRKV IRPEGIRKIV DCVDDRMLIF DPADRNPLNK VSDQVLNSMR 120
121 ARATKATASS INNSNATNKF SSQRRRHGGE IKFVFDKLFD ETSSQARVYK ETTSPLLDSV 180
181 LDGFNSTVFA YGATGCGKTY TVSGTPSQPG IIFLAMEELF NKITDLKDEK DFEISLSYLE 240
241 IYNERIRDLL KPETPSKRLV IREDTQNHIK VANLSYHHPN TVEDVMDLVV QGNINRTTSP 300
301 TEANEVSSRS HAVLQIHIMQ TNKLVDLTSQ HTFATLSIID LAGSERAAAT RNRGIRLHEG 360
361 ANINRSLLAL GNCINALCLN DGSRSCHIPY RDSKLTRLLK FSLGGNCKTV MIVCISPSSS 420
421 HYDETLNTLK YANRAKEIKT KIIRNQQSLS RHVGSYLKMI TEQKRQIEEL REREEKMISL 480
481 KLTKYKLNKE KIQLAINECV NRVQQTYAGV ETYQVAKTLK SLILCKRRFL QMVKLEVDNL 540
541 ILLFEREEST AAEMQPVISN CRMISGQLYN KIHELEMKFD ETDTLSSVIH QVHSIDLNKL 600
601 REMEDWDETY DLVYLESCLN QISELQRNEI LVNSSIMTEK LMSDPGLNSR FKFLSKWLMN 660
661 RTPNIESIIQ DLVHIDEEFE SFARTFIANP DSNFTNTNIN IINTTAADLA VPAETLQRQN 720
721 FSQKKVKWTS PDLSPSPMIE PQPELEPELH QDQDAIASEV DVSMQDTTFN EQGPSTPSAP 780
781 TTAVPRRKMR SSLLTHQSLL ATARK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
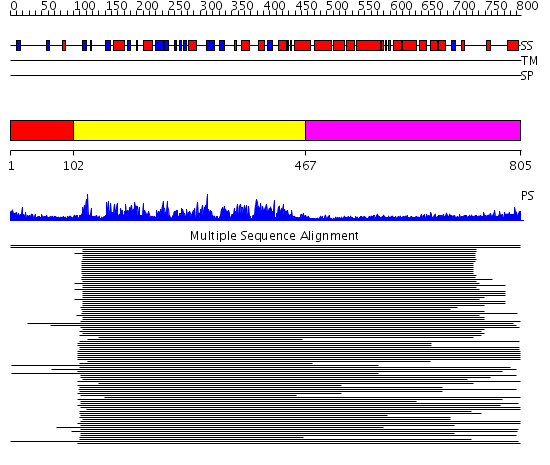
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..101] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [102..466] | 636.0103 | Kinesin | |
| 3 | View Details | [467..805] | 12.39794 | Heavy meromyosin subfragment |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)