













| General Information: |
|
| Name(s) found: |
pmk-3 /
CE29318
[WormBase]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 474 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA cytoplasm [IDA |
| Biological Process: |
MAPKKK cascade
[IGI regulation of synapse organization [IGI protein amino acid phosphorylation [IEA |
| Molecular Function: |
ATP binding
[IEA protein tyrosine kinase activity [IEA MAP kinase activity [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MASVPSSSSL PVSHVRRHED VSTPSAPPTK RSNNQSQPPE SYEPNTWLQQ QREQEQQKKL 60
61 AAENIKKQSI EATGNNEMVG EEEEDILSKP CGPHKRRFQF VMIRNITFAI PEGYDVEPNS 120
121 IEYLGGGSFG NVIKTSAVCR DGLRRYVAIK KMREPFFDPH HARRIFRETK LLQLMRHDNI 180
181 ICALDIYTPD EENDFRDVYV VTEFAGRSLY QILKQQRDYG RRVLTDEHIK FIIYQIIRAL 240
241 KYIHSANIIH RDLKPGNLAL TDDSDLMILD FGLARSLEKK DTSLTQYVQT RWYRSPEVIY 300
301 WKIDSYTNLA DMWSLGCIAA ELLTGEPLFP GDEPNAQYQR ITQLCGSPDE ELLTKIENDN 360
361 SSAIKAVIQS YTTHKRRNFR DVFSAHNPSE DFIDLLEKLL VLDPEKRITV EEAIQHPYLA 420
421 EFSLPEDEPR ADHIFDLDDS QARTRFEWRD AVWKEIMNYK RLSSSPLIPG EADR |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
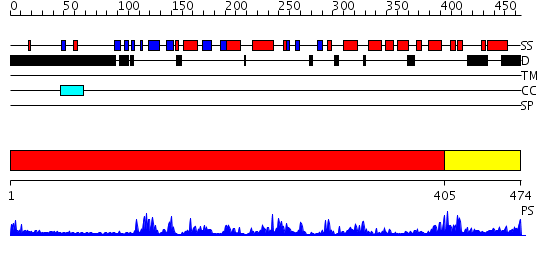
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..404] | 46.69897 | Abl tyrosine kinase, SH3 domain; Abl tyrosine kinase; Abelsone tyrosine kinase (abl) | |
| 2 | View Details | [405..474] | 89.09691 | Crystal structure of P38 with triazolopyridine |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)