













| General Information: |
|
| Name(s) found: |
MKK1 /
YOR231W
[SGD]
|
| Description(s) found:
Found 26 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae |
| Length: | 508 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cellular bud tip
[IDA |
| Biological Process: |
signal transduction
[IMP protein amino acid phosphorylation [ISS |
| Molecular Function: |
MAP kinase kinase activity
[ISS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MASLFRPPES AKCNPNSPRL KLPLLRNNQV DENNIYLTSN GSSTTAYSSH TPEPLTSSTS 60
61 TLFSQTRLHP SDSSMTLNTM KKRPAPPSLP SLSINSQSKC KTLPELVPIA DVSDGKHDLG 120
121 LKQRVIAENE LSGNSDLTPS SMASPFSHTN TSSPYLRNDL SNSVGSDFSN LISAYEQSSS 180
181 PIKSSSQPKS SSESYIDLNS VRDVDQLDEN GWKYANLKDR IETLGILGEG AGGSVSKCKL 240
241 KNGSKIFALK VINTLNTDPE YQKQIFRELQ FNRSFQSEYI VRYYGMFTDD ENSSIYIAME 300
301 YMGGRSLDAI YKNLLERGGR ISEKVLGKIA EAVLRGLSYL HEKKVIHRDI KPQNILLNEN 360
361 GQVKLCDFGV SGEAVNSLAT TFTGTSFYMA PERIQGQPYS VTSDVWSLGL TILEVANGKF 420
421 PCSSEKMAAN IAPFELLMWI LTFTPELKDE PESNIIWSPS FKSFIDYCLK KDSRERPSPR 480
481 QMINHPWIKG QMKKNVNMEK FVRKCWKD |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
| PROTEIN(S) | PUBLICATION | |
| View Data |
|
Huh WK, et al. (2003) |
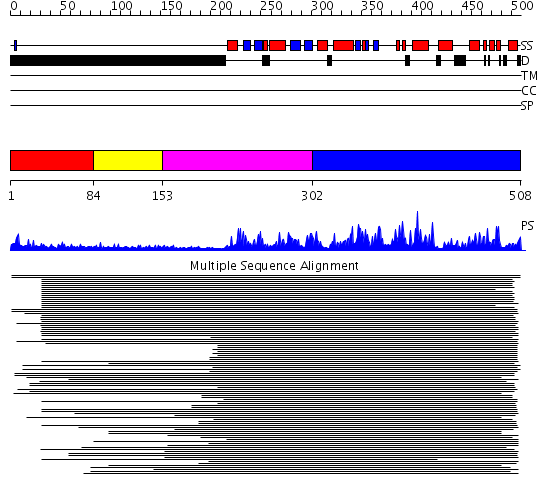
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..83] | 3.30103 | pak1 autoregulatory domain | |
| 2 | View Details | [84..152] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [153..301] | 373.9897 | pak1 | |
| 4 | View Details | [302..508] | 373.9897 | pak1 |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)