













| General Information: |
|
| Name(s) found: |
sma-4 /
CE25077
[WormBase]
|
| Description(s) found:
Found 18 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 565 amino acids |
Gene Ontology: |
|
| Cellular Component: |
intracellular
[IEA |
| Biological Process: |
dauer larval development
[IGI positive regulation of cell size [IMP nematode larval development [IMP positive regulation of organ growth [IMP tail tip morphogenesis [IGI positive regulation of protein catabolic process [IMP regulation of transcription, DNA-dependent [IEA positive regulation of multicellular organism growth [IGI |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTSQPSTSNQ MYYDPLYGAE QIVQCNPMDY HQANILCGMQ YFNNSHNRYP LLPQMPPQFT 60
61 NDHPYDFPNV PTISTLDEAS SFNGFLIPSQ PSSYNNNNIS CVFTPTPCTS SQASSQPPPT 120
121 PTVNPTPIPP NAGAVLTTAM DSCQQISHVL QCYQQGGEDS DFVRKAIESL VKKLKDKRIE 180
181 LDALITAVTS NGKQPTGCVT IQRSLDGRLQ VAGRKGVPHV VYARIWRWPK VSKNELVKLV 240
241 QCQTSSDHPD NICINPYHYE RVVSNRITSA DQSLHVENSP MKSEYLGDAG VIDSCSDWPN 300
301 TPPDNNFNGG FAPDQPQLVT PIISDIPIDL NQIYVPTPPQ LLDNWCSIIY YELDTPIGET 360
361 FKVSARDHGK VIVDGGMDPH GENEGRLCLG ALSNVHRTEA SEKARIHIGR GVELTAHADG 420
421 NISITSNCKI FVRSGYLDYT HGSEYSSKAH RFTPNESSFT VFDIRWAYMQ MLRRSRSSNE 480
481 AVRAQAAAVA GYAPMSVMPA IMPDSGVDRM RRDFCTIAIS FVKAWGDVYQ RKTIKETPCW 540
541 IEVTLHRPLQ ILDQLLKNSS QFGSS |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
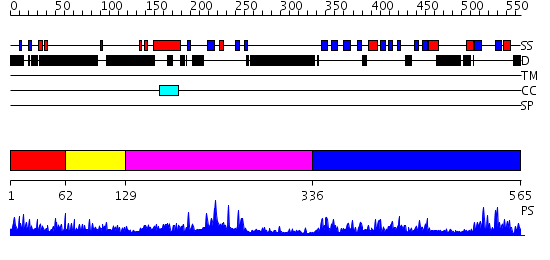
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..61] | 1.052992 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [62..128] | 1.05699 | View MSA. No confident structure predictions are available. | |
| 3 | View Details | [129..335] | 50.69897 | Crystal structure of Smad3-MH1 bound to DNA at 2.4 A resolution | |
| 4 | View Details | [336..565] | 82.09691 | Crystal Structure of the phosphorylated Smad2/Smad4 heterotrimeric complex |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.83 |
Source: Reynolds et al. (2008)