













| General Information: |
|
| Name(s) found: |
ESR1
[HGNC (HUGO)]
|
| Description(s) found:
Found 54 descriptions. SHOW ALL |
|
| Organism: | Homo sapiens |
| Length: | 595 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTMTLHTKAS GMALLHQIQG NELEPLNRPQ LKIPLERPLG EVYLDSSKPA VYNYPEGAAY 60
61 EFNAAAAANA QVYGQTGLPY GPGSEAAAFG SNGLGGFPPL NSVSPSPLML LHPPPQLSPF 120
121 LQPHGQQVPY YLENEPSGYT VREAGPPAFY RPNSDNRRQG GRERLASTND KGSMAMESAK 180
181 ETRYCAVCND YASGYHYGVW SCEGCKAFFK RSIQGHNDYM CPATNQCTID KNRRKSCQAC 240
241 RLRKCYEVGM MKGGIRKDRR GGRMLKHKRQ RDDGEGRGEV GSAGDMRAAN LWPSPLMIKR 300
301 SKKNSLALSL TADQMVSALL DAEPPILYSE YDPTRPFSEA SMMGLLTNLA DRELVHMINW 360
361 AKRVPGFVDL TLHDQVHLLE CAWLEILMIG LVWRSMEHPG KLLFAPNLLL DRNQGKCVEG 420
421 MVEIFDMLLA TSSRFRMMNL QGEEFVCLKS IILLNSGVYT FLSSTLKSLE EKDHIHRVLD 480
481 KITDTLIHLM AKAGLTLQQQ HQRLAQLLLI LSHIRHMSNK GMEHLYSMKC KNVVPLYDLL 540
541 LEMLDAHRLH APTSRGGASV EETDQSHLAT AGSTSSHSLQ KYYITGEAEG FPATV |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
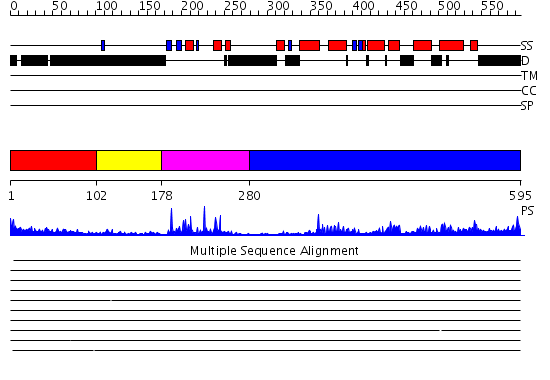
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..101] | 38.677781 | No description for PF02159.6 was found. No confident structure predictions are available. | |
| 2 | View Details | [102..177] | 0.974 | Crystal Structure of Rat Synapsin I C Domain Complexed to Ca.ATP | |
| 3 | View Details | [178..279] | 22.0 | Estrogen receptor DNA-binding domain | |
| 4 | View Details | [280..595] | 57.39794 | Estrogen receptor alpha |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.64 |
Source: Reynolds et al. (2008)