













| General Information: |
|
| Name(s) found: |
YTA12_SCHPO
[Swiss-Prot]
|
| Description(s) found:
Found 3 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe 972h- |
| Length: | 773 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MRNPFLTFRA PTRKTGDYLV SKFVKKDNFS SLRLARAYTF STRSTAVSQF SLLSLSQRSF 60
61 QSLKINKGIP EKHKIPLISS KQFSVTSKRS QNGSSGSNSD ANGRKNGQKN DDSKKKGLNG 120
121 NDPKKVFEIA LNGNTILGGI LVAYILYNVL SPNANMQEIT WQDFRQQFLD KGLVERLVVV 180
181 NRNMVRVILR GGVASGSGQY YFSIGSIDSF DRKLEDAQRQ LGIPPSEFVP VAYHDEVSVL 240
241 ATLLSFAPTL LIIGSVIYLS RRASGAAGGG QGGIFGIGKS RAKMFNHETD IKIKFADVAG 300
301 VDEAKEEIME FVKFLKNPKF YERLGAKIPR GAILSGPPGT GKTLLAKATA GEANVPFLSV 360
361 SGSEFLEMFV GVGPSRVRDL FATARKNAPC IIFIDEIDAI GKARGRGGQF GSNDERESTL 420
421 NQLLVEMDGF TSSEHIVVFA GTNRPDVLDP ALLRPGRFDR QITIDRPDIG GREQIFKVHL 480
481 KHIKAADNID LIAKRLAVLT SGFTGADIMN VCNEGALIAA RSNSNEVQMV HFEQAIERVT 540
541 AGLEKKSRVL SPEEKNTVAH HEAGHAVAGW FMEYVDPLLK VSIIPRAQAL GYASYLPKDQ 600
601 YLMSRGQILD QMGMALAGRV SEEIFFGPEK ITSGASDDFQ KVTRMAQAYV TQYGMSPTVG 660
661 TIAYPIDTRE TVQKPFSEAT AQMIDEEIRK LVKHAYERTK KLLLEHKQGL ENIAQRLLQK 720
721 EVITYNEVET ILGPRPYAYK HLNISELMRQ SEYKNDHDPR NPPIPPSPQQ PSA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
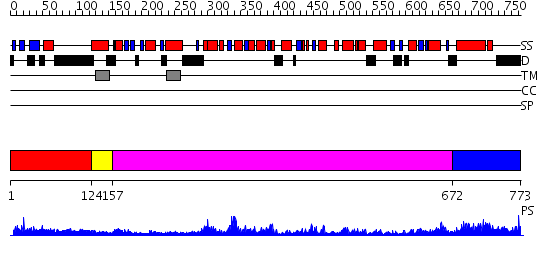
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..123] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [124..156] | 72.0 | VCP/p97 | |
| 3 | View Details | [157..671] | 25.0 | N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone; ClpA, an Hsp100 chaperone, AAA+ modules | |
| 4 | View Details | [672..773] | 114.0 | EDTA treated |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.50 |
Source: Reynolds et al. (2008)