













| General Information: |
|
| Name(s) found: |
RAD54_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 898 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MARRRLPDRP PNGIGAGERP RLVPRPINVQ DSVNRLTKPF RVPYKNTHIP PAAGRIATGS 60
61 DNIVGGRSLR KRSATVCYSG LDINADEAEY NSQDISFSQL TKRRKDALSA QRLAKDPTRL 120
121 SHIQYTLRRS FTVPIKGYVQ RHSLPLTLGM KKKITPEPRP LHDPTDEFAI VLYDPSVDGE 180
181 MIVHDTSMDN KEEESKKMIK STQEKDNINK EKNSQEERPT QRIGRHPALM TNGVRNKPLR 240
241 ELLGDSENSA ENKKKFASVP VVIDPKLAKI LRPHQVEGVR FLYRCVTGLV MKDYLEAEAF 300
301 NTSSEDPLKS DEKALTESQK TEQNNRGAYG CIMADEMGLG KTLQCIALMW TLLRQGPQGK 360
361 RLIDKCIIVC PSSLVNNWAN ELIKWLGPNT LTPLAVDGKK SSMGGGNTTV SQAIHAWAQA 420
421 QGRNIVKPVL IISYETLRRN VDQLKNCNVG LMLADEGHRL KNGDSLTFTA LDSISCPRRV 480
481 ILSGTPIQND LSEYFALLSF SNPGLLGSRA EFRKNFENPI LRGRDADATD KEITKGEAQL 540
541 QKLSTIVSKF IIRRTNDILA KYLPCKYEHV IFVNLKPLQN ELYNKLIKSR EVKKVVKGVG 600
601 GSQPLRAIGI LKKLCNHPNL LNFEDEFDDE DDLELPDDYN MPGSKARDVQ TKYSAKFSIL 660
661 ERFLHKIKTE SDDKIVLISN YTQTLDLIEK MCRYKHYSAV RLDGTMSINK RQKLVDRFND 720
721 PEGQEFIFLL SSKAGGCGIN LIGANRLILM DPDWNPAADQ QALARVWRDG QKKDCFIYRF 780
781 ISTGTIEEKI FQRQSMKMSL SSCVVDAKED VERLFSSDNL RQLFQKNENT ICETHETYHC 840
841 KRCNAQGKQL KRAPAMLYGD ATTWNHLNHD ALEKTNDHLL KNEHHYNDIS FAFQYISH |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
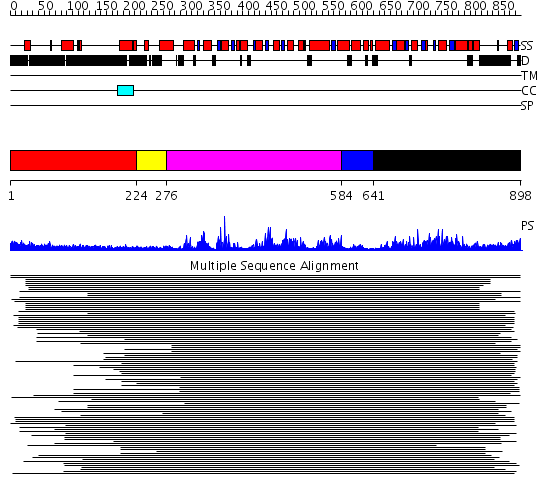
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..223] | 1.238994 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [224..275] | 13.09 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 3 | View Details | [276..583] | 13.09 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 4 | View Details | [584..640] | 8.221849 | Putative DEAD box RNA helicase | |
| 5 | View Details | [641..898] | 7.045757 | Nucleotide excision repair enzyme UvrB |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)