













| General Information: |
|
| Name(s) found: |
CHD1_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 1468 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MAAKDISTEV LQNPELYGLR RSHRAAAHQQ NYFNDSDDED DEDNIKQSRR KRMTTIEDDE 60
61 DEFEDEEGEE DSGEDEDEED FEEDDDYYGS PIKQNRSKPK SRTKSKSKSK PKSQSEKQST 120
121 VKIPTRFSNR QNKTVNYNID YSDDDLLESE DDYGSEEALS EENVHEASAN PQPEDFHGID 180
181 IVINHRLKTS LEEGKVLEKT VPDLNNCKEN YEFLIKWTDE SHLHNTWETY ESIGQVRGLK 240
241 RLDNYCKQFI IEDQQVRLDP YVTAEDIEIM DMERERRLDE FEEFHVPERI IDSQRASLED 300
301 GTSQLQYLVK WRRLNYDEAT WENATDIVKL APEQVKHFQN RENSKILPQY SSNYTSQRPR 360
361 FEKLSVQPPF IKGGELRDFQ LTGINWMAFL WSKGDNGILA DEMGLGKTVQ TVAFISWLIF 420
421 ARRQNGPHII VVPLSTMPAW LDTFEKWAPD LNCICYMGNQ KSRDTIREYE FYTNPRAKGK 480
481 KTMKFNVLLT TYEYILKDRA ELGSIKWQFM AVDEAHRLKN AESSLYESLN SFKVANRMLI 540
541 TGTPLQNNIK ELAALVNFLM PGRFTIDQEI DFENQDEEQE EYIHDLHRRI QPFILRRLKK 600
601 DVEKSLPSKT ERILRVELSD VQTEYYKNIL TKNYSALTAG AKGGHFSLLN IMNELKKASN 660
661 HPYLFDNAEE RVLQKFGDGK MTRENVLRGL IMSSGKMVLL DQLLTRLKKD GHRVLIFSQM 720
721 VRMLDILGDY LSIKGINFQR LDGTVPSAQR RISIDHFNSP DSNDFVFLLS TRAGGLGINL 780
781 MTADTVVIFD SDWNPQADLQ AMARAHRIGQ KNHVMVYRLV SKDTVEEEVL ERARKKMILE 840
841 YAIISLGVTD GNKYTKKNEP NAGELSAILK FGAGNMFTAT DNQKKLEDLN LDDVLNHAED 900
901 HVTTPDLGES HLGGEEFLKQ FEVTDYKADI DWDDIIPEEE LKKLQDEEQK RKDEEYVKEQ 960
961 LEMMNRRDNA LKKIKNSVNG DGTAANSDSD DDSTSRSSRR RARANDMDSI GESEVRALYK1020
1021 AILKFGNLKE ILDELIADGT LPVKSFEKYG ETYDEMMEAA KDCVHEEEKN RKEILEKLEK1080
1081 HATAYRAKLK SGEIKAENQP KDNPLTRLSL KKREKKAVLF NFKGVKSLNA ESLLSRVEDL1140
1141 KYLKNLINSN YKDDPLKFSL GNNTPKPVQN WSSNWTKEED EKLLIGVFKY GYGSWTQIRD1200
1201 DPFLGITDKI FLNEVHNPVA KKSASSSDTT PTPSKKGKGI TGSSKKVPGA IHLGRRVDYL1260
1261 LSFLRGGLNT KSPSADIGSK KLPTGPSKKR QRKPANHSKS MTPEITSSEP ANGPPSKRMK1320
1321 ALPKGPAALI NNTRLSPNSP TPPLKSKVSR DNGTRQSSNP SSGSAHEKEY DSMDEEDCRH1380
1381 TMSAIRTSLK RLRRGGKSLD RKEWAKILKT ELTTIGNHIE SQKGSSRKAS PEKYRKHLWS1440
1441 YSANFWPADV KSTKLMAMYD KITESQKK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
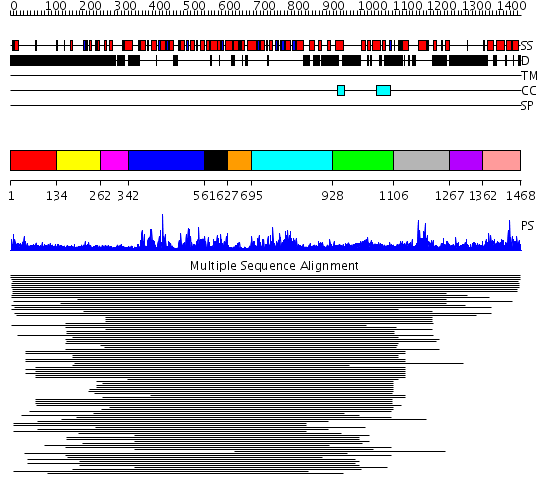
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..133] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [134..261] | 17.065502 | 'chromo' (CHRromatin Organisation MOdifier) domain No confident structure predictions are available. | |
| 3 | View Details | [262..341] | 13.61 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 4 | View Details | [342..560] | 13.61 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 5 | View Details | [561..626] | 12.85 | Putative DEAD box RNA helicase | |
| 6 | View Details | [627..694] | 7.221849 | Putative DEAD box RNA helicase | |
| 7 | View Details | [695..927] | 7.30103 | Nucleotide excision repair enzyme UvrB | |
| 8 | View Details | [928..1105] | 1.175981 | View MSA. Confident ab initio structure predictions are available. | |
| 9 | View Details | [1106..1266] | 3.051976 | View MSA. No confident structure predictions are available. | |
| 10 | View Details | [1267..1361] | N/A | No confident structure predictions are available. | |
| 11 | View Details | [1362..1468] | 1.009974 | View MSA. Confident ab initio structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 9 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 10 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 11 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)