













| General Information: |
|
| Name(s) found: |
gi|151941168
[NCBI NR]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae YJM789 |
| Length: | 757 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MASLEDLIPT VNKLQDVMYD SGIDTLDLPI LAVVGSQSSG KSSILETLVG RDFLPRGTGI 60
61 VTRRPLVLQL NNISPNSPLI EEDDNSVNPH DEVTKISGFE AGTKPLEYRG KERNHADEWG 120
121 EFLHIPGKRF YDFDDIKREI ENETARIAGK DKGISKIPIN LKVFSPHVLN LTLVDLPGIT 180
181 KVPIGEQPPD IEKQIKNLIL DYIATPNCLI LAVSPANVDL VNSESLKLAR EVDPQGKRTI 240
241 GVITKLDLMD SGTNALDILS GKMYPLKLGF VGVVNRSQQD IQLNKTVEES LDKEEDYFRK 300
301 HPVYRTISTK CGTRYLAKLL NQTLLSHIRD KLPDIKTKLN TLISQTEQEL ARYGGVGATT 360
361 NESRASLVLQ LMNKFSTNFI SSIDGTSSDI NTKELCGGAR IYYIYNNVFG NSLKSIDPTS 420
421 NLSVLDVRTA IRNSTGPRPT LFVPELAFDL LVKPQIKLLL EPSQRCVELV YEELMKICHK 480
481 CGSAELARYP KLKSMLIEVI SELLRERLQP TRSYVESLID IHRAYINTNH PNFLSATEAM 540
541 DDIMKTRRKR NQELLKSKLS QQENGQTNGI NGTSSISSNI DQDSAKNSDY DDDGIDAESK 600
601 QTKDKFLNYF FGKDKKGQPV FDASDKKRSI AGDGNIEDFR NLQISDFSLG DIDDLENAEP 660
661 PLTEREELEC ELIKRLIVSY FDIIREMIED QVPKAVMCLL VNYCKDSVQN RLVTKLYKET 720
721 LFEELLVEDQ TLAQDRELCV KSLGVYKKAA TLISNIL |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
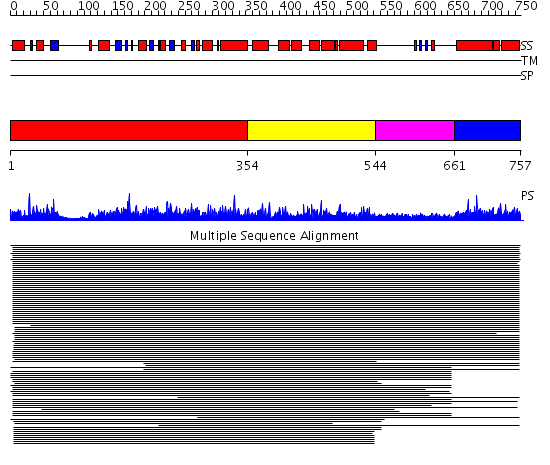
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..353] | 1175.0 | Dynamin G domain | |
| 2 | View Details | [354..543] | 50.318759 | Dynamin central region No confident structure predictions are available. | |
| 3 | View Details | [544..660] | 15.39794 | Dynamin | |
| 4 | View Details | [661..757] | 42.552842 | Dynamin GTPase effector domain Confident ab initio structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)