













| General Information: |
|
| Name(s) found: |
gi|4586029
[NCBI NR]
gi|25287692 [NCBI NR] gi|15225358 [NCBI NR] |
| Description(s) found:
Found 8 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 596 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MDSAKGWFQK RQMRGGSRYK GASGGAGGGG SNGSADEHNV ETDEEAVSNT TKQKVAAAKQ 60
61 YIENHYKEQM KILQERKERR SMLEQKLADA DVSEEDQNNL LKFLEKKETE YMRLQRHKLG 120
121 VADFDLLTMI GKGAFGEVRV CREKTTGQVY AMKKLKKAEM LRRGQVEHVR AERNLLAEVD 180
181 SNYIVKLYCS FQDDDHLYLV MEYLPGGDMM TLLMRKDTLT EEEAKFYVAE TVLAIESIHR 240
241 HNYIHRDIKP DNLLLDRYGH LRLSDFGLCK PLDCSAIGEN DFSNNSNGST EQEAGSTAPK 300
301 RTQQEQLEHW QRNRRTLAYS TVGTPDYIAP EVLLKKGYGM ECDWWSLGAI MYEMLVGYPP 360
361 FYSDDPMSTC RKIVNWKSHL KFPEEAILSR EAKDLINSLL CSVRRRLGSK GADELKAHTW 420
421 FETVDWDTIF DMDAAFVPEV NDDLDTQNFE KFDESESETQ TSSKSGPWRK MLSSKDINFV 480
481 GYTYKNFEIV NDYQVPGMGT HLLDHCGVEE EEKVDATNGQ VTLRYPTTSF TGPKFGYFHV 540
541 NLEYNGSSET SDSSETTSRP PCDRPPPAPP VVQGSFLKLL PPELEVRPKQ EGSEAC |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
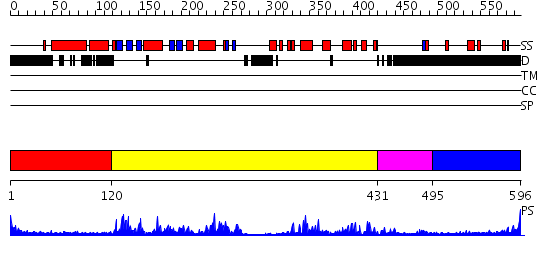
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..119] | 4.69897 | No description for 2uvmA was found. | |
| 2 | View Details | [120..430] | 51.0 | Crystal Structure Of The Human P21-Activated Kinase 5 | |
| 3 | View Details | [431..494] | 83.0 | Crystal Structure of ROCK 1 bound to fasudil | |
| 4 | View Details | [495..596] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.96 |
Source: Reynolds et al. (2008)