













| General Information: |
|
| Name(s) found: |
gi|55959718
[NCBI NR]
gi|55661763 [NCBI NR] gi|55661528 [NCBI NR] gi|55661174 [NCBI NR] gi|4758128 [NCBI NR] gi|156230969 [NCBI NR] gi|119628955 [NCBI NR] |
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Homo sapiens |
| Length: | 729 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSFGRDMELE HFDERDKAQR YSRGSRVNGL PSPTHSAHCS FYRTRTLQTL SSEKKAKKVR 60
61 FYRNGDRYFK GIVYAISPDR FRSFEALLAD LTRTLSDNVN LPQGVRTIYT IDGLKKISSL 120
121 DQLVEGESYV CGSIEPFKKL EYTKNVNPNW SVNVKTTSAS RAVSSLATAK GSPSEVRENK 180
181 DFIRPKLVTI IRSGVKPRKA VRILLNKKTA HSFEQVLTDI TDAIKLDSGV VKRLYTLDGK 240
241 QVMCLQDFFG DDDIFIACGP EKFRYQDDFL LDESECRVVK STSYTKIASS SRRSTTKSPG 300
301 PSRRSKSPAS TSSVNGTPGS QLSTPRSGKS PSPSPTSPGS LRKQRSSQHG GSSTSLASTK 360
361 VCSSMDENDG PGEEVSEEGF QIPATITERY KVGRTIGDGN FAVVKECVER STAREYALKI 420
421 IKKSKCRGKE HMIQNEVSIL RRVKHPNIVL LIEEMDVPTE LYLVMELVKG GDLFDAITST 480
481 NKYTERDASG MLYNLASAIK YLHSLNIVHR DIKPENLLVY EHQDGSKSLK LGDFGLATIV 540
541 DGPLYTVCGT PTYVAPEIIA ETGYGLKVDI WAAGVITYIL LCGFPPFRGS GDDQEVLFDQ 600
601 ILMGQVDFPS PYWDNVSDSA KELITMMLLV DVDQRFSAVQ VLEHPWVNDD GLPENEHQLS 660
661 VAGKIKKHFN TGPKPNSTAA GVSVIALDHG FTIKRSGSLD YYQQPGMYWI RPPLLIRRGR 720
721 FSDEDATRM |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
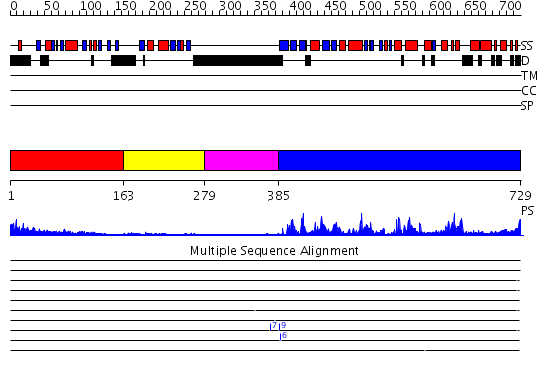
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..162] | 14.522879 | Solution structure of the N-terminal DCX domain of human doublecortin-like kinase | |
| 2 | View Details | [163..278] | 4.71 | No description for 2dnfA was found. | |
| 3 | View Details | [279..384] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [385..729] | 72.69897 | CALMODULIN-DEPENDENT PROTEIN KINASE FROM RAT |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)