













| General Information: |
|
| Name(s) found: |
tup12 /
SPAC630.14c
[Sanger Pombe]
|
| Description(s) found:
Found 16 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 586 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA nuclear chromatin [IC cytoplasm [IDA cytosol [IDA protein complex [RCA |
| Biological Process: |
chromatin remodeling in response to cation stress
[IEP negative regulation of transcription by glucose [IMP chromatin silencing [IMP response to cation stress [IMP |
| Molecular Function: |
protein binding
[IPI histone binding [IDA transcription corepressor activity [IDA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MITVRQFTFT IFKFQFMATS NVSSRVNELL EAVKKEFEDI CQKTKTVEAQ KDDFEYKAMI 60
61 SAQINEMALM KQTVMDLEMQ QSKVKDRYEE EITSLKAQLE ARRKEIASGV VPQSSKTKHG 120
121 RNSVSFGKYG NAGPFNSDNS SKPLILNNGS SGGTPKNLRS PAIDSDGTVL APIQTSNVDL 180
181 GSQYYSSPHV RPAVGATMAG SAMRTFPSNL PLGHPPPPSD SANSSVTPIA APLVVNGKVS 240
241 GNPPYPAEII PTSNVPNREE KDWTVTSNVP NKEPPISVQL LHTLEHTSVI CYVRFSADGK 300
301 FLATGCNRAA MVFNVETGKL ITLLQEESSK REGDLYVRSV AFSPDGKYLA TGVEDQQIRI 360
361 WDIAQKRVYR LLTGHEQEIY SLDFSKDGKT LVSGSGDRTV CLWDVEAGEQ KLILHTDDGV 420
421 TTVMFSPDGQ FIAAGSLDKV IRIWTSSGTL VEQLHGHEES VYSVAFSPDG KYLVSGSLDN 480
481 TIKLWELQCV SNVAPSMYKE GGICKQTFTG HKDFILSVTV SPDGKWIISG SKDRTIQFWS 540
541 PDSPHSQLTL QGHNNSVISV AVSPNGHCFA TGSGDLRARI WSYEDL |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
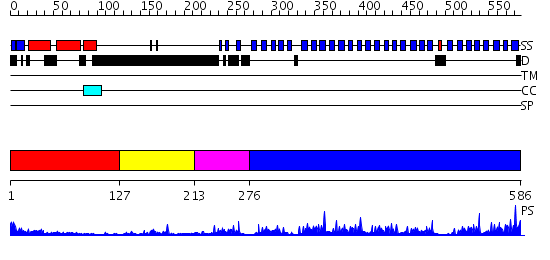
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..126] | 1.25 | Ribonuclease domain of colicin E3; Colicin E3 translocation domain; Colicin E3 receptor domain | |
| 2 | View Details | [127..212] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [213..275] | 7.0 | TolB, C-terminal domain; TolB, N-terminal domain | |
| 4 | View Details | [276..586] | 60.39794 | Tup1, C-terminal domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.96 |
Source: Reynolds et al. (2008)